| 送件注意事項 |
| 送件之樣品種類 |
| 樣品種類 |
純化方式與注意事項 |
| Colony/Culture |
請標明樣品名稱、菌種所含 Plasmid 及抗生素種類 |
| Plasmid DNA |
請標明 DNA 純化所使用 Kit 種類,並標示 DNA 濃度。DNA 濃度需大於 0.1 μg/μl,一個定序反應至少提供 10 μl。 |
| PCR Product |
PCR 產物需大於 0.05 μg/μl,體積約 10 μl,附上電泳圖照片並標明 sample 及 DNA marker 的量,並請於訂單上註明是否已純化。 |
| Primer |
一次定序反應 primer 需 5 μl,濃度應大於 5 μM。 |
|
| 送件注意事項 |
1.定序所需的 Primer 可選用本公司免費提供之 Universal Primer,並於表格上註明。
|
待測各式樣品請以宅急便以 ”冷藏方式” 運送至本公司,運費則由本公司支付。
|
3.提供樣品如為菌液,需提供 3-5ml 並且為 24 小時內新鮮之菌液,避免菌體太老或死亡,造成抽出品質不佳之 plasmid DNA。
|
4.樣品請裝於 1.5ml eppendorf tube 或 0.2 ml PCR Tube,附上 DNA template 及單一股 primer,勿將Forward 及 Reverse primer 混合。
|
5.若待分析的 DNA 定序樣品已知有特殊結構(如 GC-rich 或是 Homopolymeric regions 存在),請於送件單的備註欄註明,我們將以特殊條件做處理。
|
6.PCR 產物定序用之 primer 請標示 GC content、濃度、Tm 值(建議 primer Tm = 50°C~55°C)及 PCR時annealing 的溫度。
|
7.針對 PCR 產物如存有其他非單一產物,本公司目前尚未提供切膠服務,需由客戶自行切膠後送件,我們將提供膠體純化之服務,但需注意膠體大小需小於200mg,若膠體過太則會影響純化後 DNA 之品質。
|
8.樣品種類縮寫代碼 B-Bacterial Colony;L-Liquid Culture;D-Plasmid DNA;P-PCR Product,請勾選清楚,以方便人員作業。
|
9.若樣品為菌體或plasmid DNA,請標示清楚vector;若為PCR產物,請務必勾選未純化或已純化,若樣品已純化處理完成請標明純化方式。
|
10.若須於定序完成後歸還 plate 或純化所得之DNA樣品,請於備註欄上註明;樣品及引子保留時間為一個月。
|
11.由於定序結果皆由e-mail寄發,所以請務必正確的填上您的e-mail address。
|
12.若對實驗方法或結果有疑問,歡迎使用 e-Mail 或電話與本公司技術人員討論。
|
| 送件表格下載 |
空白定序單 |
| 定序引子列表 |
| 下表為本公司備有之常用定序引子,若您使用的引子為下表列出者,定序時可不須附上引子 ,直接使用本公司提供的高純度引子! |
| No. |
引子名稱 |
引子序列 |
| 1. |
M13F (-40) |
5'-CAG GGT TTT CCC AGT CAC GAC-3' |
| 2. |
M13R(-48) |
5'-AGC GGA TAA CAA TTT CAC ACA GG-3' |
| 3. |
M13F(-20) |
5'-GTA AAA CGA CGG CCA GT-3' |
| 4. |
M13R(-24) |
5'-AAC AGC TAT GAC CAT G-3' |
| 5. |
T7P |
5'-TAA TAC GAC TCA CTA TAG G-3' |
| 6. |
T7T |
5'-GCT AGT TAT TGC TCA GCG G-3' |
| 7. |
SP6 |
5'-ATT TAG GTG ACA CTA TAG AA-3' |
| 8. |
T3 |
5'-GCA ATT AAC CCT CAC TAA AGG G-3' |
| 9. |
BGHR |
5'-TAG AAG GCA CAG TCG AGG-3' |
| 10. |
SK |
5'-GCT CTA GAA CTA GTG GAT C-3' |
| 11. |
KS |
5'-TCG AGG TCG ACGGTATCG-3' |
| 12. |
PQEF |
5'-GGC GTA TCA CGA GGC CCT TTC G-3' |
| 13. |
PQER |
5'-CAT TAC TGG ATC TAT CAA CAG G-3' |
| 14. |
PGEX5 |
5'-GGG CTG GCA AGC CAC GTT TGG TG-3' |
| 15. |
PGEX3 |
5'-CCG GGA GCT GCA TGT GTC AGA GG-3' |
| 16. |
Nus-tag |
5'-GAA AAA GCC GGA GCA CTG ATT ATG-3' |
| 17. |
S-tag |
5'-CGA ACG CCA GCA CAT GGA CA-3' |
| 18. |
PEGFP-C5 |
5'-ATC ACA TGG TCC TGC TGG AGT TC-3' |
| 19. |
PEGFP-C3 |
5'-TTT ATG TTT CAG GTT CAG GGG-3' |
| 20. |
PEGFP-N5 |
5'-TAG GCG TGT ACG GTG GGA GG-3' |
| 21. |
PEGFP-N3 |
5'-GAC ACG CTG AAC TTG TGG CCG-3' |
| 22. |
RV primer3 |
5'-CTA GCA AAA TAG GCT GTC CC-3' |
| 23. |
RV primer4 |
5'-GAC GAT AGT CAT GCC CCG CG-3' |
| 24. |
GL2 primer |
5'-CTT TAT GTT TTT GGC GTC TTC CA-3' |
| 25. |
5'-AOX |
5'-GAC TGG TTC CAA TTG ACA AGC-3' |
| 26. |
3'-AOX |
5'-GCA AAT GGC ATT CTG ACA TCC-3' |
| 27. |
pCMV-CMV 24 |
5'-ACA AGG CTG GTG GGC ACT GGA GTG-3' |
| 28. |
CMV-taq-T7 |
5’-TAATACGACTCACTATAGGG-3’ |
| 29. |
CMV-taq-T3 |
5'-ATTAACCCTCACTAAAGGGA-3' |
| 30. |
pQE-F(2) |
5'-AAGTGCCACCTGACGTCTAAG-3' |
| 31. |
pQE-R(2) |
5'-GGAGTTCTGAGGTCATTACTG-3' |
| 32. |
PLNCX5' |
5'-AGC TCG TTT AGT GAA CCG TCA GAT C-3' |
| 33. |
PLNCX3' |
5'-ACC TAC AGG TGG GGT CTT TCA TTC CC-3' |
| 34. |
TAF |
5'-CAA GGC GAT TAA GTT GGG TA-3' |
| 35. |
TAR |
5'-TGG AAT TGT GAG CGG ATA ACA-3 |
| 36. |
u6 promoter |
5'-TACAAAATACGTGACGTAG-3' |
| 37. |
CMVIE |
5'-TGT ACG GTG GGA GGT CTA-3' |
| 38. |
PDNR-F |
5'-CAT TAT ACG AAG TTA TCA GTC GA-3 |
| 39. |
PDNR-R |
5'-AAC AGC TAT GAC CAT GTT CA-3' |
| 40. |
pCMV-CMV 30(old) |
5'-TTT CCA AAA TGT CGT AAT AAC CCC GCC CC-3' |
| 41. |
PCS-T7 |
5'-AAT ACG ACT CAC TAT AG-3' |
| 42. |
pB42ADF |
5'-CCA GCC TCT TGC TGA GTG GAG ATG-3' |
| 43. |
pB42ADR |
5'-AAG CCG ACA ACC TTG ATT GGA G-3' |
| 44. |
PBV220F |
5'-AAG AAG GGC AGC ATT CAA AG-3' |
| 45. |
PBV220R |
5'-CTG CGT TCT GAT TTA ATC TG-3' |
| 46. |
α -factor |
5'-TAC TAA TGC CAG CAT TGC TGC-3' |
| 47. |
5'AD |
5'-AAT ACC ACT ACA ATG GAT GAT G-3' |
| 48. |
3'AD |
5'-GAG ATG GTG CAC GAT GCA CAG T-3' |
| 49. |
PcDNA3.1F |
5'-CTA GAG AAC CCA CTG CTT AC-3' |
| 50. |
P17110 |
5'-TAC CAC TAC AAT GGA TG-3' |
| 51. |
P12584 |
5'-TTT TCA GTA TCT ACG AT-3' |
| 52. |
P24990 |
5'-TCA TCG GAA GAG AGT AG-3' |
| 53. |
P31430 |
5'-CGT TTT AAA ACC TAA GAG TCA C-3' |
| 54. |
F |
5'-CAT GGT ATG GCT AGC ATG AC-3' |
| 55. |
R |
5'-CAG GCT GAA AAT CTT CTC TC-3' |
| 56. |
P5' |
5'-TGC GTA CTG CGG TGA TCA AC-3' |
| 57. |
P3' |
5'-CTG CAA GGC GAT TAA GTT GG-3' |
| 58. |
S1 |
5'-CAA CGT GAA AAA ATT ATT ATT CGC-3' |
| 59. |
S6 |
5'-GTA AAT GAA TTT TCT GTA TGA GG-3' |
| 60. |
GL1 |
5'-TGT ATC TTA TGG TAC TGT AAC TG-3' |
| 61. |
Pinpoint Primer |
5'-CGT GAC GCG GTG CAG GGC G-3' |
| 62. |
3'BD |
5'-TAA GAG TCA CTT TAA AAT TTG TAT C-3' |
| 63. |
BAC1 |
5'-AAC CAT CTC GCA AAT AAA TA-3' |
| 64. |
BAC2 |
5'-ACG CAC AGA ATC TAG CGC TT-3' |
| 65. |
pCMV-F |
5'-TCTAAAAGCTGCGGAATTGT-3' |
| 66. |
pCMV-R |
5'-TCCAAACTCATCAATGTATC-3' |
| 67. |
pTRC99C-F |
5'-TTG CGC CGA CAT CAT AAC-3' |
| 68. |
pTRC99C-R |
5'-CTGCGTTCTGATTTAATCTG-3' |
|
| 定序報告圖檔判定教學 |
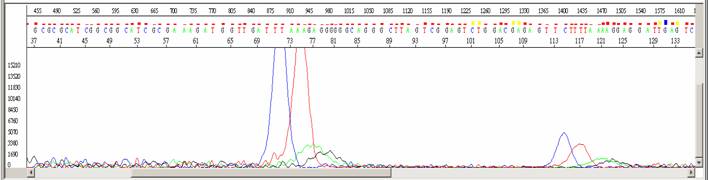
定序失敗

|
| 狀況描述 |
- ●圖形呈現雜亂無章的波峰。
- ●除了在70 bp 與 110 bp 左右有強烈的染料訊號外,其餘皆為低訊號值。
- ●平均訊號強度低於100。
- ●其訊號無法與預期的序列相符合,或者無法比對到 GeneBank 中的任何序列
|
| 發生的可能原因 |
- ●DNA 品質不佳。
- ●在酒精沉澱純化步驟時,發生 DNA lose。
- ●太少的 template DNA
- ●使用錯誤的引子。
- ●定序反應用的水受到污染。受污染的水中含有抑制反應的物質。
- ●合成引子失敗或引子降解。使用的引子在合成時失敗,所以無法用於定序。
- ●定序試劑失去活性。BigDye 試劑若儲存太久,或者儲存在非適當環境下會造成 Taq 活性喪失與螢光標定 nucleotides 的螢光衰退。
- ●毛細管阻塞。
|
| 解決方式 |
- ●DNA 品質不佳。使用純化試劑組進行二次純化,去除 DNA 中的雜質。
- ●在酒精沉澱純化步驟時,發生 DNA lose。改用市售的純化試劑組來替代酒精沉澱法,通常會有不錯的效果,相對的成本較高。
- ●太少的 template DNA。在做定序反應前,將 template DNA 跑一次 agarose gel 用以確定 DNA 濃度,並且也可以順便確定樣品是否有genomic DNA與RNA的污染。
- ●使用錯誤的引子。重新確認 primer 的序列及其正確性。
- ●定序反應用的水受到污染。請使用乾淨無污染的水。
- ●引子降解。重新合成引子,將其回溶保存於適當環境下。
- ●定序試劑失去活性。公司內部試劑皆經嚴格管理,所以這種狀況較少發生。
|
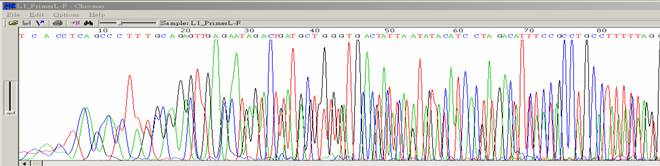
| 序列出現混合訊號 |
 |
| 狀況描述 |
- ●序列一開始會有兩個以上的波峰出現。
- ●平均訊息值正常。
|
| 發生的可能原因 |
●在定序反應時,有其他template DNA的污染。
●Primer污染。兩個以上可以與template DNA作用的primer一起做定序反應。
●PCR產物未純化乾淨,殘存forward與reverse primers。
●Template DNA 上有兩個以上的位置可以被同一個定序用的primer黏合。常見於擁有universal priming site的PCR產物接合到載體上。
●定序反應時primer annealing溫度調太低。
●在純化定序後的產物時汙染到其他樣品的產物。 |
| 解決方式 |
- ●確定template DNA 只有一個。可先跑洋菜膠電泳粗略確定是否有其他DNA污染,但是即使在膠上只看到單一產物,也無法代表沒有其他微量的非預期產物出現,或沒有其他相似大小的非預期產物在其中。
- ●確認template DNA上是否可以被同一個定序用的primer 黏合。
- ●確認在純化PCR產物時是否將殘存的 primers 及 dNTP 去除乾淨。
- ●確認定序primer的Tm值。若primer的Tm值高於定序所設定的annealing溫度5℃以上時,則調高定序時的annealing溫度。
|
| |
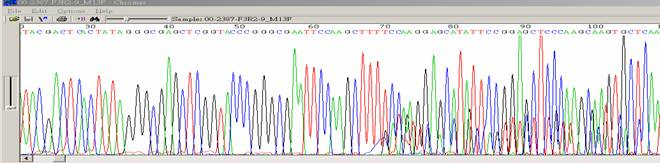 |
| 狀況描述 |
- ●同一處會有兩個以上的波峰出現。
- ●重疊序列一般出現在50bp 之後cloning site 的起始位置,較低的序列與預期的序列不同,所謂two clones。
|
| 發生的可能原因 |
- ●在定序反應時,有其他template DNA的污染。
- ●挑取single colony時同時挑到兩個colonies。常發生在兩個single colony靠太近時不小心污染到。
- ●確定template DNA 只有一個。可先跑洋菜膠電泳粗略確定是否有其他DNA污染,但是即使在膠上只看到單一產物,也無法代表沒有其他微量的非預期產物出現,或沒有其他相似大小的非預期產物在其中。
- ●避免挑取single colony時同時挑到兩個colonies。如發生此狀況建議重新挑選新的colony。
|
 |
| 狀況描述 |
- ●序列前端混亂且有明顯的終止點,後端則波峰清楚可判讀。
|
| 發生的可能原因 |
| ●一般發生於PCR 產物定序時,出現非預期的產物,干擾主要產物的定序結果。 |
| 解決方式 |
| ●重新進行純化,去除非預期產物。如果主要產物與非預期產物片段大小不同,可利用洋菜膠體進行分離後純化出單一產物。 |
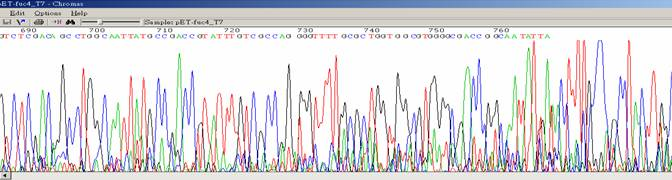
| 訊號讀不長或品質不好的訊號值 |
 |
| 狀況描述 |
- ●波峰看起來混亂且粗糙
- ●訊號在800 bp前就斷掉。
|
| 發生的可能原因 |
- ●太少template DNA。
- ●BigDye試劑稀釋太多倍。
- ●引子太多。
- ●DNA品質不佳。
- ●遇到特殊序列無法順利定序的片段。
|
| 解決方式 |
- ●確認DNA濃度是否適當。
- ●檢查BigDye稀釋的濃度是否過當。
- ●確認定序引子濃度是否正確。
- ●確認DNA 品質。
- ●試用二級結構試劑解決特殊序列問題。在定序反應時加入7-deaza-deoxy guanosine triphosphate (7-deaza-dGTP),可以幫助polymerase通過GC-rich區域。
|
|
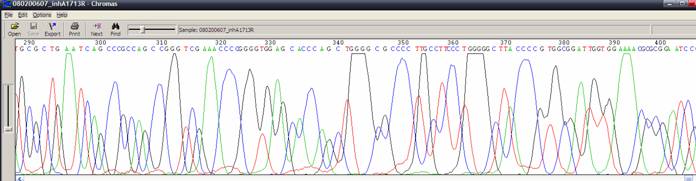 |
| |
| 狀況描述 |
- ●在500 bp前波峰呈現圓寬狀。
- ●兩個訊號之間沒有明顯的區隔。
|
| 發生的可能原因 |
- ●毛細管電泳超過負荷。樣品中殘存的鹽類與蛋白質,導致波峰解析度差,沒辦法出現尖而明顯的波峰。
- ●毛細管電泳所用的電位差(Voltage)過高。
- ●所使用緩衝液(buffer)或電泳膠(polymer) 品質不佳。
|
| 解決方式 |
- ●確認樣品是否品質良好。
- ●確認毛細管電泳所用的電位差(Voltage)。
- ●檢查所使用緩衝液(buffer)或電泳膠(polymer)是否品質穩定。
|
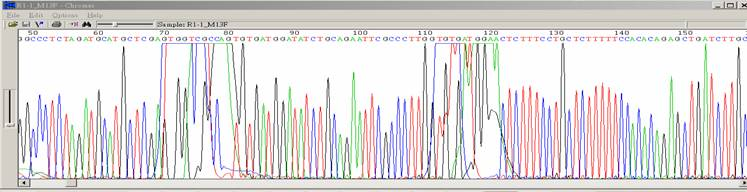
| BigDye染料殘留 |
 |
| |
| 狀況描述 |
- ●在50-70bp,110-140bp之間出現強烈的染料訊號。
|
| 發生的可能原因 |
- ●定序結束後的純化步驟不完全。
- ●定序後的產物流失。
- ●定序反應失敗。
|
| 解決方式 |
- ●確認定序產物的純化步驟是否正確。確定所使用的酒精濃度與鹽濃度是否正確,靜置時間與離心的時間是否正確。
- ●酒精沉澱中使用酒精清洗的步驟時,注意不要流失定序產物。
- ●使用其他方式取代酒精沉澱法純化產物。
|
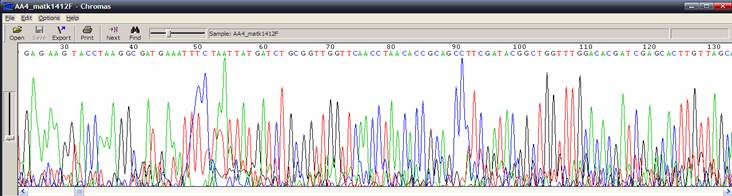
| 訊號微弱或出現雜亂背景 |
 |
| |
| 狀況描述 |
- ●在主要波峰下方出現其他微弱的波峰。
- ●在300 bp前的訊號值低於150
|
| 發生的可能原因 |
- ●定序反應失敗。
- ●太少template DNA。
- ●在純化定序產物的步驟時造成產物的流失。
- ●使用primer 黏合度不佳。
|
| 解決方式 |
| ●解決方式如同定序失敗反應的改進辦法。 |
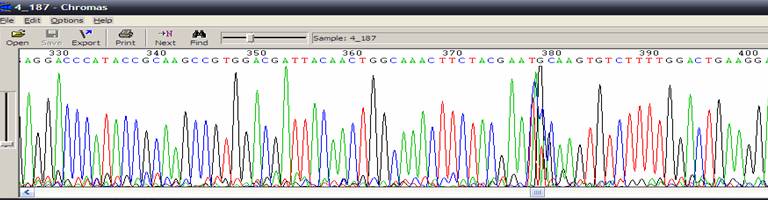
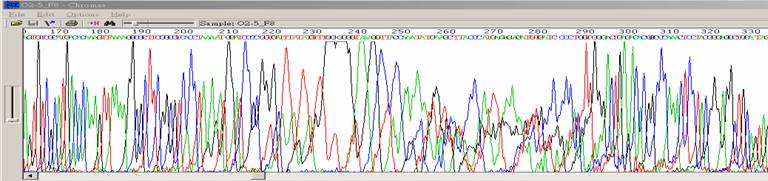
| 出現高聳且擁擠的波峰 |
|
| |
| 狀況描述 |
- ●這團波峰出現高聳狀,此高聳訊號超過能偵測的頂線值。
- 這團波峰特別高於周圍的波峰。
|
| 發生的可能原因 |
| ●毛細管電泳膠內有氣泡或雜質干擾,影響DNA泳動。 |
| 解決方式 |
| |
|
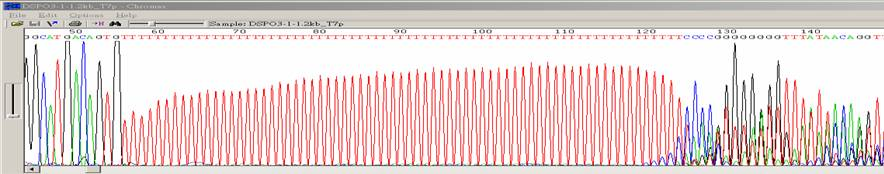
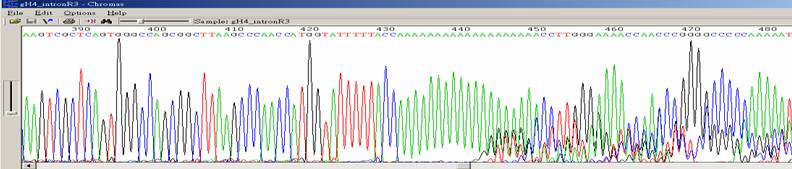
|
| 狀況描述 |
- ●在一串很長的單核酸序列後方,訊號開始雜亂。
- ●這種狀況也會發生在一連串雙核酸重複的序列後方。
|
| 發生的可能原因 |
| ●DNA在PCR中的extension步驟向外形成loop再雜交黏合後所產生。 |
| 解決方式 |
- ●以另外一端的primer再定序一次。
- 跳過連續單核酸序列的區域,另外設計primer來定序。
|
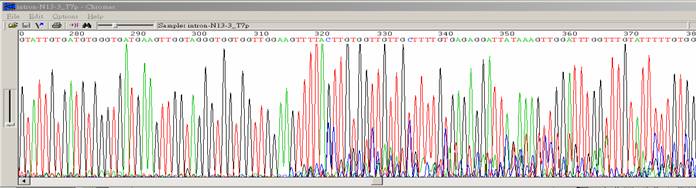
| 突然訊號中止 |


|
| 狀況描述 |
- ●訊號突然中止。
- ●一開始訊號值都不錯,突然波峰降低,訊號值變低且出現雜訊。
- ●特別是在訊號中止之前,template DNA出現G/C rich的序列。
|
| 發生的可能原因 |
- ●訊息中止的區域出現二級結構包含GC Rich、Hairpin structure、siRNA constructs、重複兩個核苷酸序列(如GT、CT)等。這個區域折疊成髮夾結構,使得Taq polymerase無法順利通過這段區域。
|
| 解決方式 |
- ●用二級結構試劑定序。在定序反應時加入7-deaza-deoxy guanosine triphosphate (7-deaza-dGTP),可以幫助polymerase通過GC-rich區域是加入DMSO,Betaine等解開二級結構的試劑,幫助您解決困難序列的定序。。
- ●使用另一端primer定序。
|
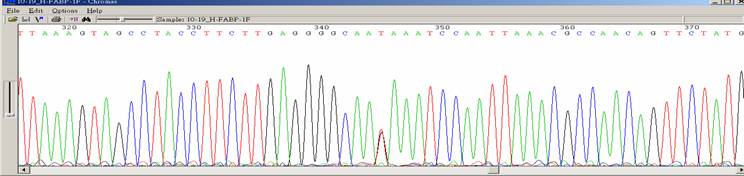
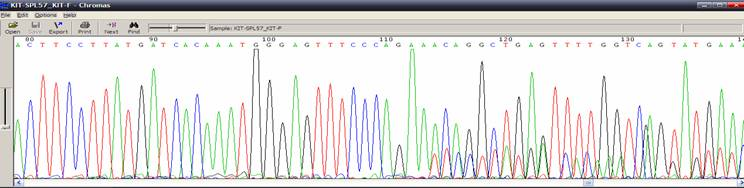
| Template DNA有突變狀況(SNP、insertion、deletion) |
|
| |
| 狀況描述 |
- ●序列中出現單一鹼基有雙重訊號。
- ●所有波峰都很乾淨,卻在某個地方出現兩個重疊波峰。
|
| 發生的可能原因 |
- ●PCR DNA發生突變。
- ●在做cloning時,DNA發生突變,特別是plasmid construct對細菌有毒性。
|
| 解決方式 |
- ●假如template DNA是heterozygous時,將PCR產物先接合到vector上再定序。
- ●假設plasmid construct對細菌有毒性,試著換個low copy number的vector,或改用其他strain的勝任細胞。
- ●用另外一端primer定序。
|
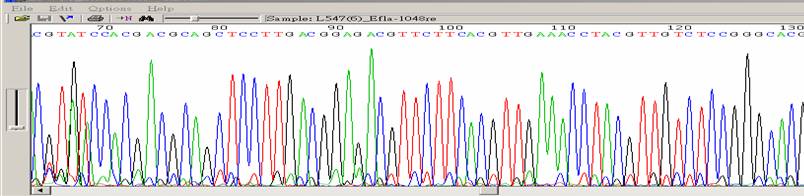
| Pirmer degraded(N-1.N-2…) |
 |
| |
| 狀況描述 |
- ●在圖形下方可以發現有較小的波峰與上面主要波峰相差一個鹼基位置,所謂shift。
|
| 發生的可能原因 |
| ●定序的 primer 發生降解,得到不同長度的 primer混和物,如此可能會導致非專一性的黏合。 |
| 解決方式 |
- ●避免重複解凍冷凍primer太多次。定序時,建議使用新鮮配製的primer。
|
| |
| 圖檔軟體下載 |
 |
| Chromas |
| version: 2.23 |
| size: 418 KB |
| Windows 95/NT4.0 or higher is required |
| Copyright © 2003-2008 Technelysium Pty Ltd |
|
- ●Opens chromatogram files from Applied Biosystems and Amersham MegaBace DNA sequencers.
- ●Opens SCF format chromatogram files created by ALF, Li-Cor, Visible Genetics OpenGene, Beckman CEQ 2000XL and CEQ 8000, and other sequencers.
- ●View Genescan genotype files.
- ●Save in SCF or Applied Biosystems format.
- ●Prints chromatogram with options to zoom or fit to one page.
- ●Exports sequences in plaint text, formatted with base numbering, FASTA, EMBL, GenBank or GCG formats.
- ●Copy the sequence to the clipboard in plain text or FASTA format for pasting into other applications.
- ●Export sequences from batches of chromatogram files, with automatic removal of vector sequence.
- ●Reverse & complement the sequence and chromatogram.
- ●Search for sequences by exact matching or optimal alignment.
- ●Display translations in 3 frames along with the sequence.
- ●Copy an image of a chromatogram section for pasting into documents or presentations.
|
|